ヘルプ
- MiFuP Safetyのしくみ
MiFuP Safetyは、微生物のゲノム情報の中から有害性に関わる遺伝子を検索し、
その微生物の有害性を推定することができるデータベースです。MiFuP Safetyにおいて、微生物の有害性機能を推定するしくみについて解説します。
- 検索対象の遺伝子について本データベースで検索できる遺伝子は、構機独自の文献調査に基づき、毒素生産能や薬剤耐性能などの有害性機能に関与する遺伝子としてリストアップした遺伝子のうち、 オーソログの検出条件(遺伝子検出条件)が設定できたものに限ります。 機構では、これらの遺伝子の検出条件やタンパク質名等の情報をまとめた遺伝子定義(NRULE)を作成し、有害性遺伝子の検索を可能にしました。
収録されている全遺伝子については遺伝子リスト で確認できます。
で確認できます。
- 遺伝子セットについて本データベースにおいては、機構が独自の文献調査に基づき、有害性機能の発揮に必要な遺伝子の組合せ等についてまとめた機能定義(NFUNC)を作成し、 検出した遺伝子から機能を推定することを可能にしています。
機能定義(NFUNC)では、以下のⅠ、Ⅱのように複数の遺伝子が必要とされる場合を含め、機能の発揮に必要な遺伝子群を「遺伝子セット」として定義しています。 有害性遺伝子を検索した結果、遺伝子セットが揃う場合に有害性機能があると推定します。
収録されている全遺伝子セットについては機能リスト で確認できます。
で確認できます。
- タンパク質が複合体を形成して機能を発揮する場合
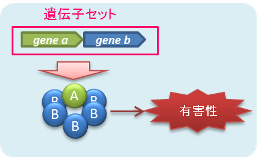 複数のタンパク質が結合し複合体を形成することで有害性機能が発揮される場合、各構成タンパク質がそれぞれ別の遺伝子にコードされているため、機能の発揮には、それらの遺伝子が揃っていることが必要となります。
複数のタンパク質が結合し複合体を形成することで有害性機能が発揮される場合、各構成タンパク質がそれぞれ別の遺伝子にコードされているため、機能の発揮には、それらの遺伝子が揃っていることが必要となります。
- 有害性機能を発揮するまでに複数の反応を経る場合
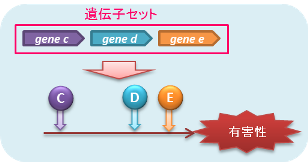 複数の酵素等が関わる一連の反応を経て有害性機能が発揮される場合、この経路に必要なタンパク質をコードする遺伝子が全て揃っていることが必要となります。
複数の酵素等が関わる一連の反応を経て有害性機能が発揮される場合、この経路に必要なタンパク質をコードする遺伝子が全て揃っていることが必要となります。
- ゲノム塩基配列から有害性機能を推定する工程についてMiFuP Safetyは下記Ⅰ~Ⅳの工程を経て、微生物のゲノム塩基配列から有害性機能を推定します。
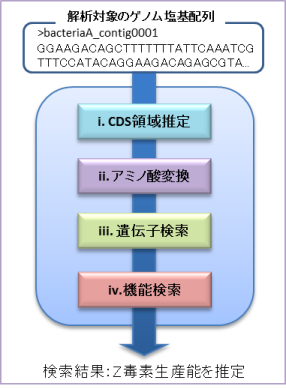
- ゲノム塩基配列からCDS(coding sequence)領域を推定
- 塩基配列をアミノ酸配列に変換細菌・植物プラスチドのコドン表[no.11]を用いて、CDS領域の塩基配列をアミノ酸配列に変換します。
- 遺伝子検索Ⅱで得られたアミノ酸配列から、MiFuP Safetyで定義した有害性遺伝子のオーソログを検出するための条件(遺伝子検出条件)に当てはまるものを検索します。 遺伝子検出条件は、以下のいずれか、又は組み合わせで設定しています。
- BLAST検索のスコア値(Score)と同一性(Identities)の閾値
閾値はオーソログの値を考慮して、遺伝子毎に設定しています。 - ドメインやモチーフ
InterPro Scan(タンパク質のファミリー・ドメイン・モチーフに関する統合データベース)の検索結果をもとに、 配列中のドメインやモチーフ等の存在を評価します。
 で確認できます。
で確認できます。
- BLAST検索のスコア値(Score)と同一性(Identities)の閾値
- 機能検索
- MiFuP Safetyのしくみ
MiFuP Safetyは、微生物のゲノム情報の中から有害性に関わる遺伝子を検索し、
その微生物の有害性を推定することができるデータベースです。MiFuP Safetyにおいて、微生物の有害性機能を推定するしくみについて解説します。
- 検索対象の遺伝子について本データベースで検索できる遺伝子は、構機独自の文献調査に基づき、毒素生産能や薬剤耐性能などの有害性機能に関与する遺伝子としてリストアップした遺伝子のうち、 オーソログの検出条件(遺伝子検出条件)が設定できたものに限ります。 機構では、これらの遺伝子の検出条件やタンパク質名等の情報をまとめた遺伝子定義(NRULE)を作成し、有害性遺伝子の検索を可能にしました。
収録されている全遺伝子については遺伝子リスト で確認できます。
で確認できます。
- 遺伝子セットについて本データベースにおいては、機構が独自の文献調査に基づき、有害性機能の発揮に必要な遺伝子の組合せ等についてまとめた機能定義(NFUNC)を作成し、 検出した遺伝子から機能を推定することを可能にしています。
機能定義(NFUNC)では、以下のⅠ、Ⅱのように複数の遺伝子が必要とされる場合を含め、機能の発揮に必要な遺伝子群を「遺伝子セット」として定義しています。 有害性遺伝子を検索した結果、遺伝子セットが揃う場合に有害性機能があると推定します。
収録されている全遺伝子セットについては機能リスト で確認できます。
で確認できます。
- タンパク質が複合体を形成して機能を発揮する場合
 複数のタンパク質が結合し複合体を形成することで有害性機能が発揮される場合、各構成タンパク質がそれぞれ別の遺伝子にコードされているため、機能の発揮には、それらの遺伝子が揃っていることが必要となります。
複数のタンパク質が結合し複合体を形成することで有害性機能が発揮される場合、各構成タンパク質がそれぞれ別の遺伝子にコードされているため、機能の発揮には、それらの遺伝子が揃っていることが必要となります。
- 有害性機能を発揮するまでに複数の反応を経る場合
 複数の酵素等が関わる一連の反応を経て有害性機能が発揮される場合、この経路に必要なタンパク質をコードする遺伝子が全て揃っていることが必要となります。
複数の酵素等が関わる一連の反応を経て有害性機能が発揮される場合、この経路に必要なタンパク質をコードする遺伝子が全て揃っていることが必要となります。
- ゲノム塩基配列から有害性機能を推定する工程についてMiFuP Safetyは下記Ⅰ~Ⅳの工程を経て、微生物のゲノム塩基配列から有害性機能を推定します。

- ゲノム塩基配列からCDS(coding sequence)領域を推定
- 塩基配列をアミノ酸配列に変換細菌・植物プラスチドのコドン表[no.11]を用いて、CDS領域の塩基配列をアミノ酸配列に変換します。
- 遺伝子検索Ⅱで得られたアミノ酸配列から、MiFuP Safetyで定義した有害性遺伝子のオーソログを検出するための条件(遺伝子検出条件)に当てはまるものを検索します。 遺伝子検出条件は、以下のいずれか、又は組み合わせで設定しています。
- BLAST検索のスコア値(Score)と同一性(Identities)の閾値
閾値はオーソログの値を考慮して、遺伝子毎に設定しています。 - ドメインやモチーフ
InterPro Scan(タンパク質のファミリー・ドメイン・モチーフに関する統合データベース)の検索結果をもとに、 配列中のドメインやモチーフ等の存在を評価します。
 で確認できます。
で確認できます。
- BLAST検索のスコア値(Score)と同一性(Identities)の閾値
- 機能検索
- 機能検索
微生物ゲノムの塩基配列・アミノ酸配列から有害性に関わる遺伝子を検索し、微生物の有害性機能を推定します。
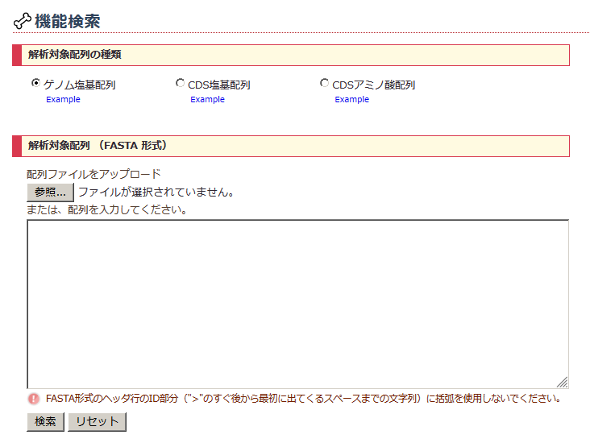
- 検索の仕方
-
解析をおこなう配列の種類を「解析対象配列の種類」を選択してください。
-
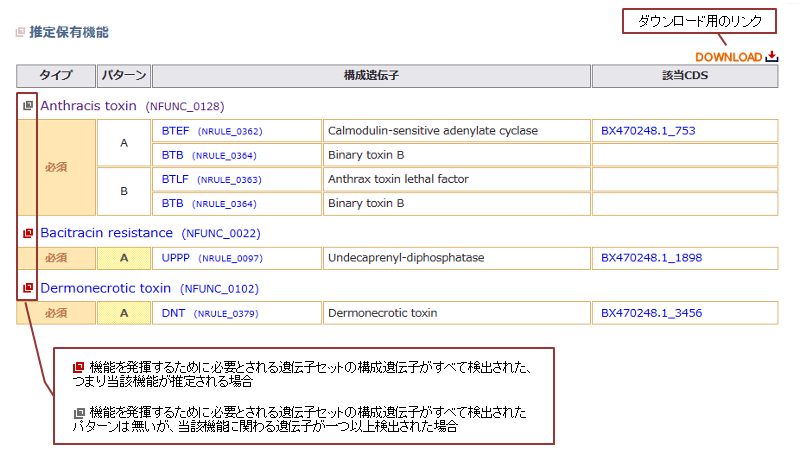
- 検索結果ページMiFuP Safetyで解析対象配列(クエリ配列)について機能検索した結果、推定された機能について、機能名、その構成遺伝子、クエリCDS名を表示します。
- 有害性機能を発揮するために必要な遺伝子セット
 の構成遺伝子がすべて検出された機能のみ表示します。
の構成遺伝子がすべて検出された機能のみ表示します。 - 遺伝子セットの構成遺伝子のうち一部の遺伝子のみが検出された場合は、検出された遺伝子と欠けている遺伝子の情報を「検索結果詳細ページ」に表示します。
ダウンロード用のリンクから、検索結果(CSV形式)及びクエリCDS名の塩基配列とアミノ酸配列(multi-FASTA形式)がダウンロードできます。
<画面表示項目の説明> 項目名 説明  (機能)
(機能)クエリ配列から推定された機能の名前です。MiFuP Safetyでつけた機能名です。(リンク先:機能情報ページ) タイプ 機能発揮に必要とされる遺伝子を”必須”、必須ではないが関与する遺伝子を”任意”と表示します。 パターン 機能発揮に必須な構成遺伝子セットのパターン名です。遺伝子セットが1つしかない場合はパターン名「A」が表示されます。 遺伝子セットのパターンが複数存在する場合は、各パターンに対応するアルファベット1文字のパターン名が表示されます。 構成遺伝子 遺伝子セットのパターンを構成する遺伝子です。左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。 (リンク先:遺伝子情報ページ) クエリCDS名 有害性遺伝子として検出されたクエリ配列CDSの名前です。ゲノム塩基配列をクエリとした場合は、Prodigal(gene-finding program)が付与したCDS IDが表示されます。 クリックすると、検出されたクエリCDSのアミノ酸配列が表示されます。
実際の表示画面(例)
- 有害性機能を発揮するために必要な遺伝子セット
- 検索結果詳細ページ
- 検索結果の補足説明
- 遺伝子セットのパターンが複数存在する場合有害性機能を発揮するために必要な遺伝子セット
 のパターンが複数ある場合、
機能検索では、遺伝子セットのいずれかのパターンについて、構成遺伝子がすべて検出された場合に、その機能があると推定します。
のパターンが複数ある場合、
機能検索では、遺伝子セットのいずれかのパターンについて、構成遺伝子がすべて検出された場合に、その機能があると推定します。
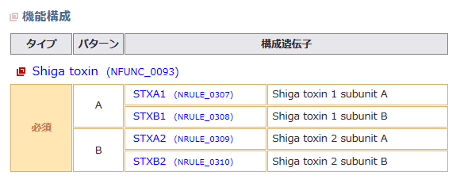
- 機能が推定されなかった場合について機能検索した結果、機能が推定されなかった場合、検索結果ページには以下の2つのいずれかが表示されます。
- 遺伝子セット
 の構成遺伝子のうち、一部の遺伝子のみが検出された場合下図のとおり、検索結果詳細ページへのリンクが表示されます。
の構成遺伝子のうち、一部の遺伝子のみが検出された場合下図のとおり、検索結果詳細ページへのリンクが表示されます。
検索結果詳細ページでは、検出された構成遺伝子の情報が確認できます。 ヒットしたCDS配列のヘッダ名(塩基配列を入力した場合はgene finding 後自動的に予測された各遺伝子領域へ割り振られた名)は「クエリCDS名」欄に表示されます。 また検出されなかった遺伝子については、「クエリCDS名」が空欄となります。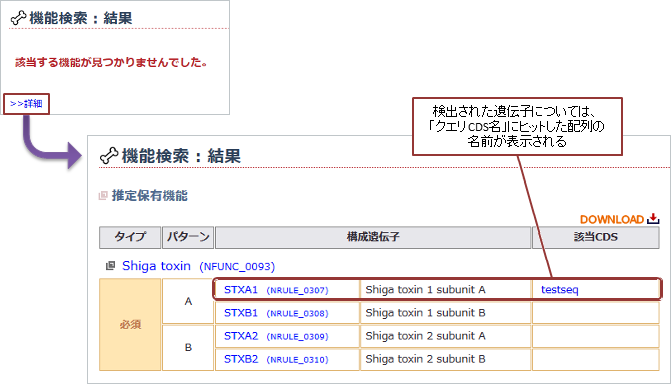
- 遺伝子が検出されなかった場合検索結果ページは下図のとおりです。検索結果詳細ページへのリンクは表示されません。

- リスト
リストページでは、MiFuP Safetyで検索できる機能及び遺伝子の一覧を表示します。
- 機能リストMiFuP Safetyで検索できる機能一覧を、機能名(英名)のアルファベット順に表示します。
各列のタイトルをクリックするとソートされます。
検索窓では絞り込み検索をすることができます。
機能一覧はCSV形式でダウンロードできます。画面表示項目の説明 項目名 説明 機能(英名) MiFuP Safetyでつけた機能名(英名)です。(リンク先:機能情報ページ) 機能(和名) MiFuP Safetyでつけた機能名(和名)です。(リンク先:Noteページ) ID 機能定義のIDです。 MiFuP Safety 固有のIDであり、「“NFUNC_”+4桁の番号」の形式でつけています。(リンク先:機能情報ページ) 実際の表示画面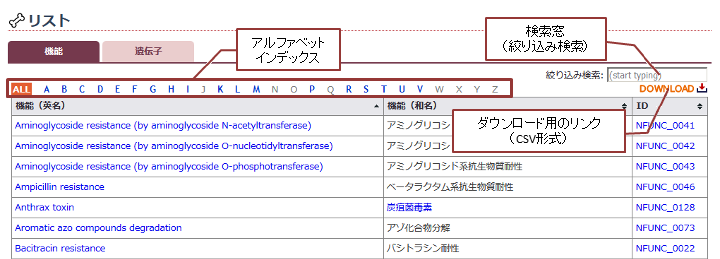
- 遺伝子リストMiFuP Safetyで検索可能な有害性に関わる遺伝子のリストを、遺伝子IDのアルファベット順に表示します。
各列のタイトルをクリックするとソートされます。
検索窓では絞り込み検索をすることができます。
遺伝子リストはCSV形式でダウンロードできます。画面表示項目の説明 項目名 説明 遺伝子ID 遺伝子のIDです。 MiFuP Safety 固有のIDであり、遺伝子名等をもとにつけています。(リンク先:遺伝子情報ページ) ID 遺伝子定義のIDです。 MiFuP Safety 固有のIDであり、「“NRULE_”+4桁の番号」の形式でつけています。(リンク先:遺伝子情報ページ) タンパク質名 この遺伝子にコードされているタンパク質の名称です。 EC IUBMBが定義した酵素番号です。 遺伝子名 一般的な遺伝子名です。 関与する機能 この遺伝子が関与する機能のIDです。(リンク先:機能情報ページ) 実際の表示画面
- サイト内検索
サイト内検索では、MiFuP Safetyサイト内の機能情報ページ、遺伝子情報ページ、Noteに対して検索できます。
キーワードを入力してください。


結果は以下の分類別に表示されます。
| 分類 | 検索対象 |
|---|---|
| 機能 | 機能定義情報。概要、構成遺伝子など。…… 機能情報ページ  |
| 遺伝子 | 遺伝子定義情報。タンパク質名、関与する機能など。…… 遺伝子情報ページ  |
| Note | 微生物の有害性機能についての情報。…… Note  |
実際の表示画面(例:「Exfoliative」を検索した場合)

- Note
MiFuP Safety Noteは、微生物の有害性機能について、そのメカニズム、関連遺伝子、文献情報をまとめたページです。
毒素の多くについては細菌毒素ハンドブック(サイエンスフォーラム社 発行)を参照にして記述しました。
毒素の多くについては細菌毒素ハンドブック(サイエンスフォーラム社 発行)を参照にして記述しました。
閲覧したい項目の機能名をクリックしてください。
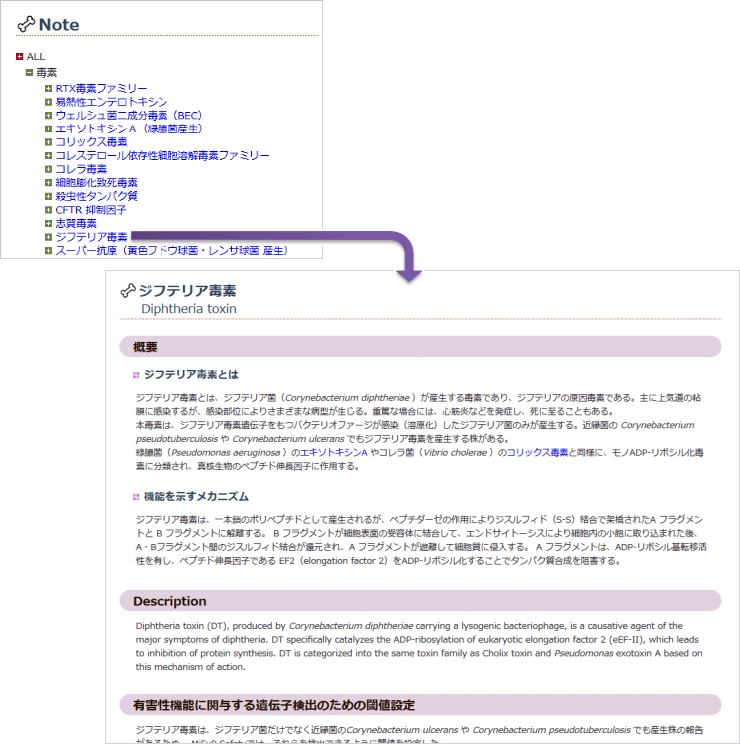
実際の表示画面(例: CFTR 抑制因子)

- 機能検索
微生物ゲノムの塩基配列・アミノ酸配列から有害性に関わる遺伝子を検索し、微生物の有害性機能を推定します。
- 検索の仕方
-
解析をおこなう配列の種類を「解析対象配列の種類」を選択してください。
-
- 検索結果ページMiFuP Safetyで解析対象配列(クエリ配列)について機能検索した結果、推定された機能について、機能名、その構成遺伝子、クエリCDS名を表示します。
- 有害性機能を発揮するために必要な遺伝子セット
 の構成遺伝子がすべて検出された機能のみ表示します。
の構成遺伝子がすべて検出された機能のみ表示します。 - 遺伝子セットの構成遺伝子のうち一部の遺伝子のみが検出された場合は、検出された遺伝子と欠けている遺伝子の情報を「検索結果詳細ページ」に表示します。
ダウンロード用のリンクから、検索結果(CSV形式)及びクエリCDS名の塩基配列とアミノ酸配列(multi-FASTA形式)がダウンロードできます。
<画面表示項目の説明> 項目名 説明  (機能)
(機能)クエリ配列から推定された機能の名前です。MiFuP Safetyでつけた機能名です。(リンク先:機能情報ページ) タイプ 機能発揮に必要とされる遺伝子を”必須”、必須ではないが関与する遺伝子を”任意”と表示します。 パターン 機能発揮に必須な構成遺伝子セットのパターン名です。遺伝子セットが1つしかない場合はパターン名「A」が表示されます。 遺伝子セットのパターンが複数存在する場合は、各パターンに対応するアルファベット1文字のパターン名が表示されます。 構成遺伝子 遺伝子セットのパターンを構成する遺伝子です。左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。 (リンク先:遺伝子情報ページ) クエリCDS名 有害性遺伝子として検出されたクエリ配列CDSの名前です。ゲノム塩基配列をクエリとした場合は、Prodigal(gene-finding program)が付与したCDS IDが表示されます。 クリックすると、検出されたクエリCDSのアミノ酸配列が表示されます。
実際の表示画面(例)
- 有害性機能を発揮するために必要な遺伝子セット
- 検索結果詳細ページ
- 検索結果の補足説明
- 遺伝子セットのパターンが複数存在する場合有害性機能を発揮するために必要な遺伝子セット
 のパターンが複数ある場合、
機能検索では、遺伝子セットのいずれかのパターンについて、構成遺伝子がすべて検出された場合に、その機能があると推定します。
のパターンが複数ある場合、
機能検索では、遺伝子セットのいずれかのパターンについて、構成遺伝子がすべて検出された場合に、その機能があると推定します。

- 機能が推定されなかった場合について機能検索した結果、機能が推定されなかった場合、検索結果ページには以下の2つのいずれかが表示されます。
- 遺伝子セット
 の構成遺伝子のうち、一部の遺伝子のみが検出された場合下図のとおり、検索結果詳細ページへのリンクが表示されます。
の構成遺伝子のうち、一部の遺伝子のみが検出された場合下図のとおり、検索結果詳細ページへのリンクが表示されます。
検索結果詳細ページでは、検出された構成遺伝子の情報が確認できます。 ヒットしたCDS配列のヘッダ名(塩基配列を入力した場合はgene finding 後自動的に予測された各遺伝子領域へ割り振られた名)は「クエリCDS名」欄に表示されます。 また検出されなかった遺伝子については、「クエリCDS名」が空欄となります。
- 遺伝子が検出されなかった場合検索結果ページは下図のとおりです。検索結果詳細ページへのリンクは表示されません。

- リスト
リストページでは、MiFuP Safetyで検索できる機能及び遺伝子の一覧を表示します。
- 機能リストMiFuP Safetyで検索できる機能一覧を、機能名(英名)のアルファベット順に表示します。
各列のタイトルをクリックするとソートされます。
検索窓では絞り込み検索をすることができます。
機能一覧はCSV形式でダウンロードできます。画面表示項目の説明 項目名 説明 機能(英名) MiFuP Safetyでつけた機能名(英名)です。(リンク先:機能情報ページ) 機能(和名) MiFuP Safetyでつけた機能名(和名)です。(リンク先:Noteページ) ID 機能定義のIDです。 MiFuP Safety 固有のIDであり、「“NFUNC_”+4桁の番号」の形式でつけています。(リンク先:機能情報ページ) 実際の表示画面
- 遺伝子リストMiFuP Safetyで検索可能な有害性に関わる遺伝子のリストを、遺伝子IDのアルファベット順に表示します。
各列のタイトルをクリックするとソートされます。
検索窓では絞り込み検索をすることができます。
遺伝子リストはCSV形式でダウンロードできます。画面表示項目の説明 項目名 説明 遺伝子ID 遺伝子のIDです。 MiFuP Safety 固有のIDであり、遺伝子名等をもとにつけています。(リンク先:遺伝子情報ページ) ID 遺伝子定義のIDです。 MiFuP Safety 固有のIDであり、「“NRULE_”+4桁の番号」の形式でつけています。(リンク先:遺伝子情報ページ) タンパク質名 この遺伝子にコードされているタンパク質の名称です。 EC IUBMBが定義した酵素番号です。 遺伝子名 一般的な遺伝子名です。 関与する機能 この遺伝子が関与する機能のIDです。(リンク先:機能情報ページ) 実際の表示画面
- サイト内検索
サイト内検索では、MiFuP Safetyサイト内の機能情報ページ、遺伝子情報ページ、Noteに対して検索できます。
キーワードを入力してください。


結果は以下の分類別に表示されます。
| 分類 | 検索対象 |
|---|---|
| 機能 | 機能定義情報。概要、構成遺伝子など。…… 機能情報ページ  |
| 遺伝子 | 遺伝子定義情報。タンパク質名、関与する機能など。…… 遺伝子情報ページ  |
| Note | 微生物の有害性機能についての情報。…… Note  |
実際の表示画面(例:「Exfoliative」を検索した場合)

- Note
MiFuP Safety Noteは、微生物の有害性機能について、そのメカニズム、関連遺伝子、文献情報をまとめたページです。
毒素の多くについては細菌毒素ハンドブック(サイエンスフォーラム社 発行)を参照にして記述しました。
毒素の多くについては細菌毒素ハンドブック(サイエンスフォーラム社 発行)を参照にして記述しました。
閲覧したい項目の機能名をクリックしてください。

実際の表示画面(例: CFTR 抑制因子)

- 機能情報ページ
MiFuP Safetyで検索できる各機能についての詳細情報です。機能名はMiFuP Safetyがつけたものです。
検索結果ページ や機能リスト
や機能リスト  に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
検索結果ページ
 や機能リスト
や機能リスト  に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
<画面表示項目の説明>
| ●機能情報 | |
| 項目名 | 説明 |
|---|---|
| 機能(和名) | MiFuP Safetyでつけた機能名(和名)です。 |
| ID | 機能定義のIDです。 MiFuP Safety 固有のIDであり、「“NFUNC_”+4桁の番号」の形式でつけています。 |
| 最終更新日 | 機能の定義の最終更新日です。 |
| 説明 |
機能概要です。詳細な説明はNote  を参照してください。 を参照してください。
|
| コメント | 作成した定義についての補足説明です。 |
| 参照 | 機能や経路などの関連情報(データベースなど)へのリンクです。 |
| ●機能構成 | |
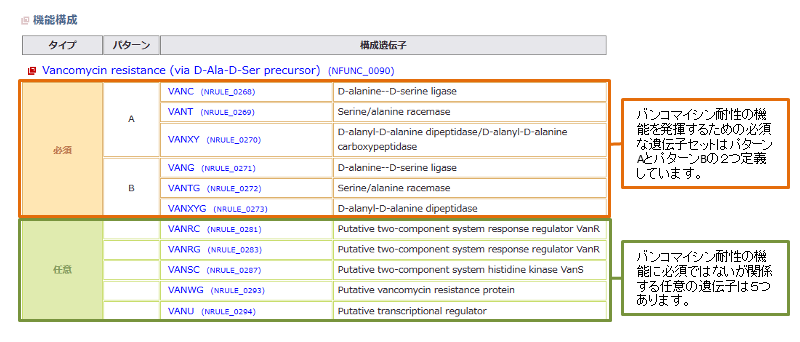
| 項目名 | 説明 |
| タイプ | 機能発揮に必要とされる遺伝子を”必須”、必須ではないが関与する遺伝子を”任意”と表示します。 |
| パターン | 機能発揮に必須な構成遺伝子セットのパターン名です。 遺伝子セットが1つしかない場合はパターン名「A」が表示されます。遺伝子セットのパターンが複数存在する場合は、各パターンに対し、 Aから順にアルファベット1文字のパターン名が付与されます。 |
| 構成遺伝子 |
遺伝子セット  のパターンを構成する遺伝子です。
左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。(リンク先:遺伝子情報ページ) のパターンを構成する遺伝子です。
左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。(リンク先:遺伝子情報ページ)
|
実際の表示画面
●機能情報(例:ボツリヌス毒素)
●機能構成(例:バンコマイシン耐性)
- 遺伝子情報ページ
MiFuP Safetyで定義づけした各遺伝子についての詳細情報です。
検索結果ページ や遺伝子リスト
や遺伝子リスト  に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
検索結果ページ
 や遺伝子リスト
や遺伝子リスト  に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
- 遺伝子情報ページ<画面表示項目の説明>
●機能情報 項目名 説明 ID 遺伝子定義のIDです。 MiFuP Safety 固有のIDであり、「“NRULE_”+4桁の番号」の形式でつけています。 遺伝子ID 遺伝子のIDです。 MiFuP Safety 固有のIDであり、遺伝子名等をもとにつけています。 最終更新日 遺伝子の定義の最終更新日です。 タンパク質名 遺伝子にコードされているタンパク質の名称です。 遺伝子名 一般的な遺伝子名です。 遺伝子条件 MiFuP Safetyで定義した、遺伝子を検出するための条件です。条件の見方については、「遺伝子条件」について  をご参照ください。
をご参照ください。
参照 遺伝子オントロジー、モチーフなど、他のデータベースの参照情報です。 配列の特徴 代表的な遺伝子配列の、活性中心等の重要な位置情報を伴うアミノ酸配列情報です。 配列のUniProtIDと微生物名を掲載しています。 注釈 その他の関連情報です。 参考文献 代表的な遺伝子配列についての参考文献です。 配列のUniProtIDと微生物名、文献のPubMedIDを掲載しています。 ●関与する機能 項目名 説明 機能(英名) この遺伝子が関与する機能の名前(英名)です。(リンク先:機能情報ページ) 機能(和名) この遺伝子が関与する機能の名前(和名)です。(リンク先:機能情報ページ) 実際の表示画面(例:Botulinum toxin type G)
- 「遺伝子条件」について有害性遺伝子の検出条件は、BLAST検索のスコア値(Score)と同一性(Identities)、ドメインやモチーフ等の有無のいずれか、又はその組み合わせで設定しています。 設定した検出条件は、遺伝子情報ページの「遺伝子条件」に以下のように掲載しています。
- BLASTP( protein-protein BLAST )の値を検出条件としている場合<条件掲載形式>BLASTP; [クエリ配列のID]; hit=[ヒット数]; score=[スコア値]; pid=[同一性]
クエリ配列のID: クエリ配列となる有害性遺伝子のUniProtのアクセッション番号。 ヒット数: クエリ配列と検索対象配列が、スコア値および同一性の条件を満たしてヒットする箇所の数の下限値。 スコア値: rawスコアの下限値。 同一性: %identityの下限値。 <掲載例>BLASTP; P10845; hit=1; score=1290; pid=80解説: UniProtのアクセッション番号P10845の配列をクエリとしてBLASTPを実行した結果、検索対象の配列に1箇所以上ヒットし、そのスコア値が1290より大きく、かつidentityが80%より大きい場合、検出対象になります。 - ドメインやモチーフ等を検出条件としている場合<条件掲載形式>[データベース名];[検出条件のID]; hit=[ヒット数]<掲載例>PFAM; PF06442; hit=1
解説: InterPro Scan で検索した結果、検索対象の配列にPfamのアクセッション番号PF06442が1箇所以上ヒットする場合、検出対象になります。 - 複数条件を組み合わせて検出条件としている場合遺伝子の検出条件が複数ある場合、それらの条件をANDもしくはORでつないで掲載しています。<条件掲載形式1>[条件1]AND[条件2]AND[条件3] ・・・
条件: Ⅰ. BLASTPとⅡ. ドメインやモチーフの各条件。 AND: 複数の条件を必要とする場合、それらの条件をANDでつないで掲載。 <掲載例1>PFAM; PF03945; hit=1 AND PFAM; PF03944; hit=1解説: 検索対象の配列にPfamのアクセッション番号PF03945が1か所以上ヒットし、かつPfamのアクセッション番号PF03944が1箇所以上ヒットする場合、検出対象になります。 <条件掲載形式2>[条件1]OR[条件2]OR[条件3]条件: Ⅰ. BLASTPとⅡ. ドメインやモチーフの各条件。 OR: 複数の条件のうち、いずれかを満たせば良い場合、それらの条件をORでつないで掲載。 <掲載例2>BLASTP; P20173; hit=1; score=630; pid=95 ①ORBLASTP; P13956; hit=1; score=600; pid=50 ②ORBLASTP; P07287; hit=1; score=850; pid=53 ③解説: ①UniProtのアクセッション番号P20173の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が630より大きく、かつidentityが95%より大きい場合、もしくは②UniProtのアクセッション番号P13956の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が600より大きく、かつidentityが50%より大きい場合、もしくは③UniProtのアクセッション番号P07287の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が850より大きく、かつidentityが53%より大きい場合、検出対象になります。
- 機能情報ページ
MiFuP Safetyで検索できる各機能についての詳細情報です。機能名はMiFuP Safetyがつけたものです。
検索結果ページ や機能リスト
や機能リスト  に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
検索結果ページ
 や機能リスト
や機能リスト  に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「機能名」又は“NFUNC_”で始まるIDをクリックすると、本ページが表示されます。
<画面表示項目の説明>
| ●機能情報 | |
| 項目名 | 説明 |
|---|---|
| 機能(和名) | MiFuP Safetyでつけた機能名(和名)です。 |
| ID | 機能定義のIDです。 MiFuP Safety 固有のIDであり、「“NFUNC_”+4桁の番号」の形式でつけています。 |
| 最終更新日 | 機能の定義の最終更新日です。 |
| 説明 |
機能概要です。詳細な説明はNote  を参照してください。 を参照してください。
|
| コメント | 作成した定義についての補足説明です。 |
| 参照 | 機能や経路などの関連情報(データベースなど)へのリンクです。 |
| ●機能構成 | |
| 項目名 | 説明 |
| タイプ | 機能発揮に必要とされる遺伝子を”必須”、必須ではないが関与する遺伝子を”任意”と表示します。 |
| パターン | 機能発揮に必須な構成遺伝子セットのパターン名です。 遺伝子セットが1つしかない場合はパターン名「A」が表示されます。遺伝子セットのパターンが複数存在する場合は、各パターンに対し、 Aから順にアルファベット1文字のパターン名が付与されます。 |
| 構成遺伝子 |
遺伝子セット  のパターンを構成する遺伝子です。
左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。(リンク先:遺伝子情報ページ) のパターンを構成する遺伝子です。
左側の項目はMiFuP SafetyでつけたIDです。右側の項目は、遺伝子にコードされているタンパク質の名称です。(リンク先:遺伝子情報ページ)
|
実際の表示画面
●機能情報(例:ボツリヌス毒素)
●機能構成(例:バンコマイシン耐性)

- 遺伝子情報ページ
MiFuP Safetyで定義づけした各遺伝子についての詳細情報です。
検索結果ページ や遺伝子リスト
や遺伝子リスト  に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
検索結果ページ
 や遺伝子リスト
や遺伝子リスト  に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
に表示される「遺伝子ID」又は“NRULE_”で始まるIDをクリックすると、本ページが表示されます。
- 遺伝子情報ページ<画面表示項目の説明>
●機能情報 項目名 説明 ID 遺伝子定義のIDです。 MiFuP Safety 固有のIDであり、「“NRULE_”+4桁の番号」の形式でつけています。 遺伝子ID 遺伝子のIDです。 MiFuP Safety 固有のIDであり、遺伝子名等をもとにつけています。 最終更新日 遺伝子の定義の最終更新日です。 タンパク質名 遺伝子にコードされているタンパク質の名称です。 遺伝子名 一般的な遺伝子名です。 遺伝子条件 MiFuP Safetyで定義した、遺伝子を検出するための条件です。条件の見方については、「遺伝子条件」について  をご参照ください。
をご参照ください。
参照 遺伝子オントロジー、モチーフなど、他のデータベースの参照情報です。 配列の特徴 代表的な遺伝子配列の、活性中心等の重要な位置情報を伴うアミノ酸配列情報です。 配列のUniProtIDと微生物名を掲載しています。 注釈 その他の関連情報です。 参考文献 代表的な遺伝子配列についての参考文献です。 配列のUniProtIDと微生物名、文献のPubMedIDを掲載しています。 ●関与する機能 項目名 説明 機能(英名) この遺伝子が関与する機能の名前(英名)です。(リンク先:機能情報ページ) 機能(和名) この遺伝子が関与する機能の名前(和名)です。(リンク先:機能情報ページ) 実際の表示画面(例:Botulinum toxin type G)
- 「遺伝子条件」について有害性遺伝子の検出条件は、BLAST検索のスコア値(Score)と同一性(Identities)、ドメインやモチーフ等の有無のいずれか、又はその組み合わせで設定しています。 設定した検出条件は、遺伝子情報ページの「遺伝子条件」に以下のように掲載しています。
- BLASTP( protein-protein BLAST )の値を検出条件としている場合<条件掲載形式>BLASTP; [クエリ配列のID]; hit=[ヒット数]; score=[スコア値]; pid=[同一性]
クエリ配列のID: クエリ配列となる有害性遺伝子のUniProtのアクセッション番号。 ヒット数: クエリ配列と検索対象配列が、スコア値および同一性の条件を満たしてヒットする箇所の数の下限値。 スコア値: rawスコアの下限値。 同一性: %identityの下限値。 <掲載例>BLASTP; P10845; hit=1; score=1290; pid=80解説: UniProtのアクセッション番号P10845の配列をクエリとしてBLASTPを実行した結果、検索対象の配列に1箇所以上ヒットし、そのスコア値が1290より大きく、かつidentityが80%より大きい場合、検出対象になります。 - ドメインやモチーフ等を検出条件としている場合<条件掲載形式>[データベース名];[検出条件のID]; hit=[ヒット数]<掲載例>PFAM; PF06442; hit=1
解説: InterPro Scan で検索した結果、検索対象の配列にPfamのアクセッション番号PF06442が1箇所以上ヒットする場合、検出対象になります。 - 複数条件を組み合わせて検出条件としている場合遺伝子の検出条件が複数ある場合、それらの条件をANDもしくはORでつないで掲載しています。<条件掲載形式1>[条件1]AND[条件2]AND[条件3] ・・・
条件: Ⅰ. BLASTPとⅡ. ドメインやモチーフの各条件。 AND: 複数の条件を必要とする場合、それらの条件をANDでつないで掲載。 <掲載例1>PFAM; PF03945; hit=1 AND PFAM; PF03944; hit=1解説: 検索対象の配列にPfamのアクセッション番号PF03945が1か所以上ヒットし、かつPfamのアクセッション番号PF03944が1箇所以上ヒットする場合、検出対象になります。 <条件掲載形式2>[条件1]OR[条件2]OR[条件3]条件: Ⅰ. BLASTPとⅡ. ドメインやモチーフの各条件。 OR: 複数の条件のうち、いずれかを満たせば良い場合、それらの条件をORでつないで掲載。 <掲載例2>BLASTP; P20173; hit=1; score=630; pid=95 ①ORBLASTP; P13956; hit=1; score=600; pid=50 ②ORBLASTP; P07287; hit=1; score=850; pid=53 ③解説: ①UniProtのアクセッション番号P20173の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が630より大きく、かつidentityが95%より大きい場合、もしくは②UniProtのアクセッション番号P13956の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が600より大きく、かつidentityが50%より大きい場合、もしくは③UniProtのアクセッション番号P07287の配列をクエリとしてBLASTPを実行した結果、 検索対象の配列に1箇所以上ヒットし、そのスコア値が850より大きく、かつidentityが53%より大きい場合、検出対象になります。