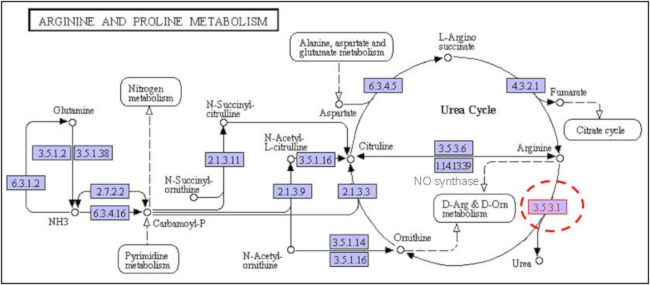
Arginine and Proline metabolism
--KEGG Pathway: map00330
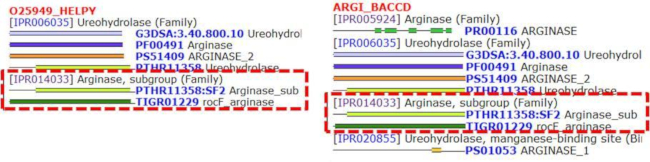
Protein structures of alginase.
O25949_HELPY , arginase of Helicobacter pylori; ARGI_BACCD
, arginase of Helicobacter pylori; ARGI_BACCD , arginase of Bacillus caldovelox
, arginase of Bacillus caldovelox
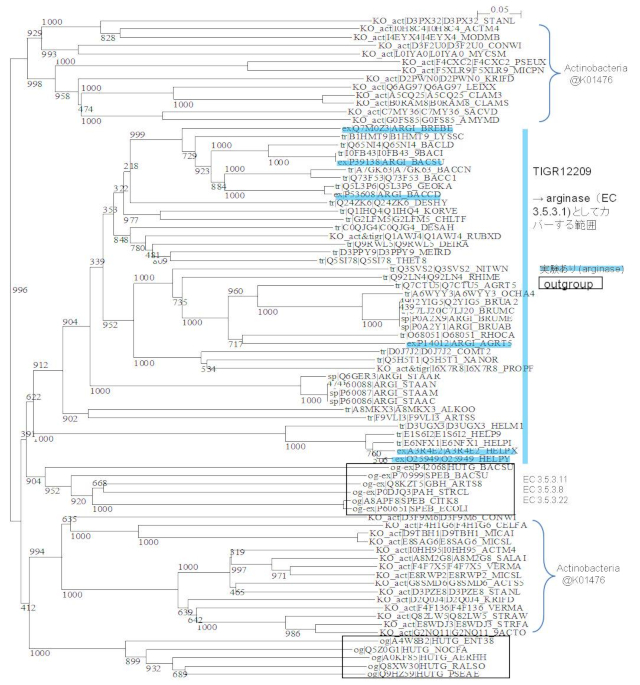
Phylogenetic tree of bacteria arginase.

アルギニン分解
概要
アルギニンは塩基性アミノ酸の一種で、蛋白質を構成するアミノ酸としては最も塩基性が高い。
機能に関する知見
機能を示すメカニズム
L-アルギニンはアルギナーゼによって分解される。アルギナーゼは尿素回路を構成する酵素の一つで、L-アルギニンを加水分解して尿素とオルニチンを生成する。
EC 3.5.3.1 arginase
arginase
Reaction: L-arginine + H2O = L-ornithine + urea
 arginase
arginaseReaction: L-arginine + H2O = L-ornithine + urea

--KEGG Pathway: map00330
一般にアルギナーゼは至適pH が 9.0-11.0 と高く、補酵素に Mn2+ を必要とするが、Helicobacter属のアルギナーゼは至適pH が 6.1 と他種の同酵素に比べて著しく低く、
Mn2+ よりも Co2+ 存在下で至適活性を示すなど異なる生化学的特徴を有する。これは酸性度の高い胃内環境に適応した結果と言われている (PMID: 21636918 )。
このような違いがあるものの、実験で活性が確認されているバクテリアのアルギナーゼは、みな同じ Arginase family (IPR014033
)。
このような違いがあるものの、実験で活性が確認されているバクテリアのアルギナーゼは、みな同じ Arginase family (IPR014033 ) に属している。
) に属している。
 )。
このような違いがあるものの、実験で活性が確認されているバクテリアのアルギナーゼは、みな同じ Arginase family (IPR014033
)。
このような違いがあるものの、実験で活性が確認されているバクテリアのアルギナーゼは、みな同じ Arginase family (IPR014033 ) に属している。
) に属している。

O25949_HELPY
 , arginase of Helicobacter pylori; ARGI_BACCD
, arginase of Helicobacter pylori; ARGI_BACCD , arginase of Bacillus caldovelox
, arginase of Bacillus caldovelox
K01476 arginase にアサインされるエントリーは、IPR014033
arginase にアサインされるエントリーは、IPR014033 arginase family のメンバーと概ねオーバーラップするが、IPR014033
arginase family のメンバーと概ねオーバーラップするが、IPR014033 には放線菌が含まれない点で異なる。
放線菌のアルギナーゼに関する実験情報は現時点では見当たらず、系統解析の結果、実験情報のあるアルギナーゼとは全く別のグループを形成したことからも、
ルール作成においては K01476
には放線菌が含まれない点で異なる。
放線菌のアルギナーゼに関する実験情報は現時点では見当たらず、系統解析の結果、実験情報のあるアルギナーゼとは全く別のグループを形成したことからも、
ルール作成においては K01476 を使用しなかった。
を使用しなかった。
 arginase にアサインされるエントリーは、IPR014033
arginase にアサインされるエントリーは、IPR014033 arginase family のメンバーと概ねオーバーラップするが、IPR014033
arginase family のメンバーと概ねオーバーラップするが、IPR014033 には放線菌が含まれない点で異なる。
放線菌のアルギナーゼに関する実験情報は現時点では見当たらず、系統解析の結果、実験情報のあるアルギナーゼとは全く別のグループを形成したことからも、
ルール作成においては K01476
には放線菌が含まれない点で異なる。
放線菌のアルギナーゼに関する実験情報は現時点では見当たらず、系統解析の結果、実験情報のあるアルギナーゼとは全く別のグループを形成したことからも、
ルール作成においては K01476 を使用しなかった。
を使用しなかった。

機能を持つことが知られている生物
Helicobacter pylori、Bacillus caldovelox、Brevibacillus brevis、Agrobacterium tumefaciens などでアルギナーゼ活性が確認されている。
実用化例
揮発しにくいアミノ酸をガスクロマトグラフィーで分析するには、クロロギ酸アルキル処理などにより誘導体化してから分析を行う必要がある。
ところがアルギニンは前処理で誘導体化した場合でも揮発しにくいため、ガスクロマトグラフィーによる分析が不可能とされていた。
試料をアルギニン分解酵素で処理し、生成物であるオルニチンを分析する手法の実用化により、迅速で安価なアミノ酸分析の提供が期待される。
参考文献
- Zhang, J. et al. (2011). Crystallization and preliminary crystallographic studies of Helicobacter pylori arginase. Acta Crystallogr Sect F Struct Biol Cryst Commun. 67(Pt 6):707-709. PMID: 21636918

- Bewley, M. C. et al. (1996). The cloning, expression and crystallisation of a thermostable arginase. FEBS Lett. 386(2-3):215-218. PMID: 8647285

関連外部リンク
- KEGG Pathway: map00330 Arginine and proline metabolism
MiFuPへのリンク
- NFUNC_0074 Arginine degradation
NRULE_0239 Arginase
(更新日 2014/03/12)