Chlor_00200 : CDS information
 Location
Location
| Organism | Amycolatopsis orientalis |
|---|---|
| Strain | A82846UV37B |
| Entry name | Chloroeremomycin |
| Contig | AJ223998 |
| Start / Stop / Direction | 46,005 / 47,747 / + [in whole cluster] 11,761 / 13,503 / + [in contig] |
| Location | 46005..47747 [in whole cluster] 11761..13503 [in contig] |
| Type | CDS |
| Length | 1,743 bp (580 aa) |
 Annotation
Annotation
| Category | 1.4 Other |
|---|---|
| Product | putative L-tyrosine--PCP ligase/peptidyl carrier protein putative L-tyrosyl-PCP synthetase/peptidyl carrier protein |
| Product (GenBank) | PCZA361.18 |
| Gene | cepK ORF19 |
| Gene (GenBank) | |
| EC number | |
| Keyword |
|
| Note | |
| Note (GenBank) |
|
| Reference |
|
| Related Reference |
|
 PKS/NRPS Module
PKS/NRPS Module
| B | 1 | 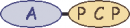 | tyrosine |
|---|
 Sequence
Sequence
>putative L-tyrosine--PCP ligase/peptidyl carrier protein [PCZA361.18] MTGEAQPPSTAPALFEATAAAVPDRPAVAMGTTTLTYAELNGEANLLAHRLVEHGVGPEK LVALAMPRSVEFVIAILAVHKAGAAYVPVDPDYPEDRRRHMLDDTSAHCLLALPGQDVAG APVVMSVERQAGLSAPNLTDQDRLGPLLPDHPAYVIYTSGSTGQPKGVLVTHRGIPNLAD DYVRRQNLVPDSRLLAFASPSFDAAVAEFWPIWLAGGCLVLAPASDLIPGEPLARLVRDQ RITHVTLPPSALAPLEEGGGLPPGLTLLVAGEACPAPVAKSWARDRVMINAYGPTEATVA VTASDPLTGEGTPPIGRPITGVSTYILDDRLHPVPDGDVGELYMTGPGLARGYLRRPAAT AERFLPNPFGGPGERMYRTGDRVWAGSDGQLVFVGRVDDQLKVRGHRIEPGEVESALLAV DGVAQAVVTEHDNRLVAYVVGAGGARVAAEDLLPPLRKQLPGYLVPDVVVGLPRLPTSPN GKVDRAALPDPDADGAERATSGRAPSTPTEIRLAELFAEVLSVSSVGVDDSFFDIGGHSL LATRLVARIRESLQVRLRVQAFFNAPSVAQLAKVLDGAST
>putative L-tyrosine--PCP ligase/peptidyl carrier protein [PCZA361.18] ATGACCGGCGAGGCACAGCCGCCATCCACTGCGCCCGCGTTGTTCGAGGCGACCGCCGCC GCGGTGCCGGACCGGCCGGCCGTGGCGATGGGAACCACCACGCTGACCTACGCAGAGCTG AACGGGGAGGCCAATCTCCTCGCGCACAGGCTCGTCGAGCACGGGGTGGGCCCGGAAAAG CTTGTCGCACTGGCGATGCCGCGGTCGGTCGAGTTCGTCATCGCGATCCTGGCCGTGCAC AAGGCAGGTGCCGCGTACGTGCCCGTCGACCCGGACTATCCGGAGGACCGCAGGCGGCAC ATGCTCGACGACACCTCAGCGCATTGTCTTTTGGCCCTGCCCGGGCAGGATGTGGCCGGT GCGCCGGTTGTGATGAGCGTGGAACGTCAGGCAGGCCTGTCCGCGCCGAACCTGACCGAC CAGGACCGGCTCGGCCCGTTGCTTCCCGATCACCCCGCGTACGTCATCTACACGTCGGGC TCGACTGGGCAGCCGAAAGGCGTACTGGTCACGCACCGCGGCATCCCGAACCTGGCCGAC GACTACGTGCGCCGCCAGAACCTGGTGCCGGACAGCAGGTTGCTGGCTTTCGCGTCACCT AGCTTCGACGCCGCCGTCGCCGAGTTCTGGCCGATCTGGCTGGCCGGTGGCTGCCTGGTG CTTGCTCCCGCGTCGGACCTGATCCCCGGCGAGCCGCTCGCCCGGCTGGTGCGCGACCAG CGGATCACCCACGTCACGTTGCCGCCGTCCGCACTGGCACCGCTCGAGGAAGGCGGCGGC CTGCCCCCGGGACTGACCCTCTTGGTCGCGGGCGAGGCGTGCCCGGCTCCGGTCGCGAAA AGCTGGGCACGCGACCGCGTCATGATCAACGCGTACGGCCCGACCGAGGCCACGGTCGCG GTGACCGCGAGCGACCCGCTGACCGGCGAAGGCACGCCGCCGATCGGCAGGCCGATCACC GGCGTGAGCACGTACATCCTGGACGACCGGCTGCACCCCGTTCCGGACGGGGACGTGGGC GAGCTGTACATGACCGGCCCTGGCCTTGCCCGCGGTTACCTGCGCAGACCGGCCGCGACC GCGGAGCGGTTCCTGCCGAATCCCTTCGGTGGCCCGGGTGAGCGCATGTACCGCACCGGC GACCGGGTGTGGGCGGGTTCGGACGGGCAGCTCGTGTTCGTCGGCCGCGTCGACGACCAG CTGAAGGTTCGTGGTCACCGGATCGAGCCGGGCGAGGTCGAGTCCGCGCTGCTCGCGGTG GATGGCGTGGCCCAGGCGGTGGTGACCGAGCACGACAACCGGCTCGTCGCTTATGTGGTG GGCGCCGGCGGGGCGCGGGTGGCAGCCGAAGACCTCCTGCCGCCGCTGCGCAAGCAGCTG CCCGGCTACCTCGTACCGGACGTGGTCGTCGGCTTGCCGCGCCTGCCGACGTCGCCGAAC GGCAAGGTCGACCGGGCCGCCTTGCCCGATCCCGATGCGGACGGCGCCGAGCGCGCGACC TCGGGGCGAGCACCGAGCACACCGACCGAGATCCGCCTGGCCGAGTTGTTCGCCGAAGTG CTCAGCGTCAGCAGCGTCGGCGTCGACGACAGCTTCTTCGACATCGGCGGCCACTCGCTG CTCGCGACTCGGCTGGTGGCCCGCATCCGGGAAAGCCTGCAGGTCCGGCTGCGCGTGCAG GCCTTCTTCAACGCGCCGTCCGTGGCACAGCTGGCCAAGGTGCTCGACGGCGCATCGACG TGA
 Feature
Feature
| BLASTP Database:UniProtKB:2011_09 |
|
|---|---|
| InterPro Database:interpro:38.0 |
IPR000873 AMP-dependent synthetase/ligase (Domain)
IPR006162 Phosphopantetheine attachment site (PTM)
IPR009081 Acyl carrier protein-like (Domain)
IPR010071 Amino acid adenylation domain (Domain)
IPR020459 AMP-binding (Domain)
IPR020845 AMP-binding, conserved site (Conserved_site)
|
| SignalP | No significant hit |
| TMHMM | No significant hit |


 Chlor_00190
Chlor_00190


