ベタイン生合成
概要
ベタイン (betaine, trimethylglycine, glycine-betaine) は好塩菌が生合成する適合溶質 (compatible solute) の代表的物質で、植物から細菌まで多くの生物体内に存在する。
高浸透圧下においてベタインを細胞内に蓄積する機能が浸透圧耐性につながることが報告されている。
機能に関する知見
機能を示すメカニズム
高浸透圧環境下において、親水性の高いベタインを細胞内に蓄積することで細胞内の水分を維持し、タンパク質などの高分子を浸透圧ストレスから保護する。
細菌のベタイン生合成は、コリンからの経路とグリシンからの経路が報告されている。
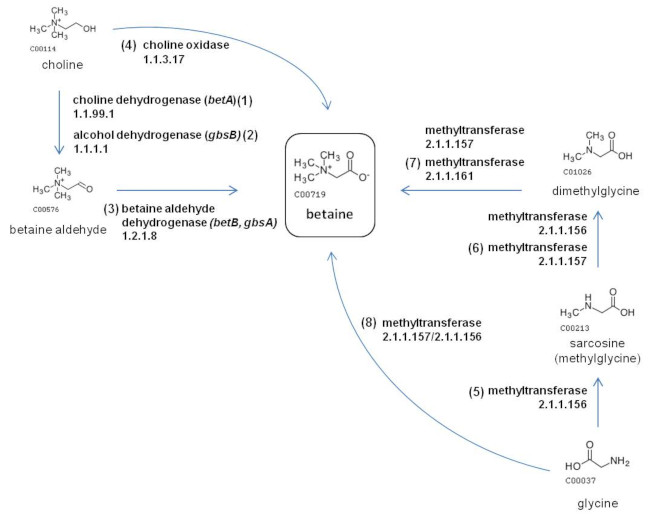
Bacterial betaine biosynthesis pathways

コリンからベタイン生合成する経路 (NFUNC_0049)
コリンから合成する経路には、betaine-aldehyde を介し、2ステップでベタインを合成する経路 (1) (2) (3) と、choline oxidase により 1ステップでベタインを合成する経路 (4) の 2つがある。
-
2ステップでベタインを合成する経路 (1) (2) (3)
コリンを betaine aldehyde に変換する酵素は生物種により異なり、Proteobacteriaや Staphylococcus 属では choline dehydrogenase BetA (EC 1.1.99.1
 , NRULE_0175 だが Bacillus 属 では gbsB にコードされている
alcohol dehydrogenase (EC 1.1.1.1
, NRULE_0175 だが Bacillus 属 では gbsB にコードされている
alcohol dehydrogenase (EC 1.1.1.1 , NRULE_0176) である。
BetA は betaine aldehyde も基質とすることができる、との実験報告があることから (PMID: 3512525
, NRULE_0176) である。
BetA は betaine aldehyde も基質とすることができる、との実験報告があることから (PMID: 3512525 , PMID: 12676692
, PMID: 12676692 ) 、
BetA を保存する種ではベタイン合成に関して betaine aldehyde dehydrogenase BetB は必須ではない。
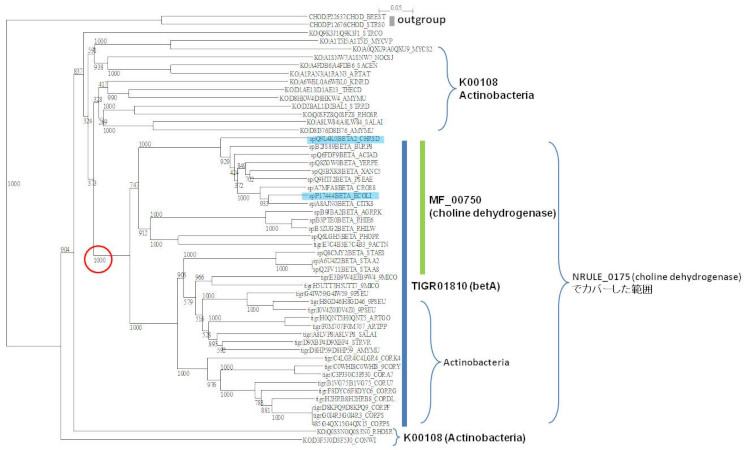
放線菌の一部では BetA に相同性を示す choline dehydrogenase が保存されている。
EC 1.1.99.1TIGR01810
) 、
BetA を保存する種ではベタイン合成に関して betaine aldehyde dehydrogenase BetB は必須ではない。
放線菌の一部では BetA に相同性を示す choline dehydrogenase が保存されている。
EC 1.1.99.1TIGR01810 choline dehydrogenase (betA) (NRULE_0175)
choline dehydrogenase (betA) (NRULE_0175) を用いて実験情報から活性があると推測される Proteobacteria、Firmicutes のエントリーを抽出することが可能である。
この EC番号の KO K00108
を用いて実験情報から活性があると推測される Proteobacteria、Firmicutes のエントリーを抽出することが可能である。
この EC番号の KO K00108 も存在するが、TIGR01810
も存在するが、TIGR01810 とはActinobacteria の種が異なっている。
K00108
とはActinobacteria の種が異なっている。
K00108 の Actinobacteria もカバーするべきかどうか系統解析で検討し、抽出条件は TIGR01810
の Actinobacteria もカバーするべきかどうか系統解析で検討し、抽出条件は TIGR01810 のみとした。
のみとした。
Phylogenetic tree of choline dehydrogenase EC 1.2.1.8TIGR01804 betaine aldehyde dehydrogenase (NRULE_0177)
betaine aldehyde dehydrogenase (NRULE_0177) で実験情報から活性があると推測される Proteobacteria、Firmicutes のエントリーは抽出可能だが、Actiniobacteria は含まない。
K00130
で実験情報から活性があると推測される Proteobacteria、Firmicutes のエントリーは抽出可能だが、Actiniobacteria は含まない。
K00130 は Actinobacteria を含むが、これらのエントリーを系統解析すると GbsA といえないものが多かった。
Actinobacteria については BLASTP で GbsA と相同性のあるもののみカバーした。
は Actinobacteria を含むが、これらのエントリーを系統解析すると GbsA といえないものが多かった。
Actinobacteria については BLASTP で GbsA と相同性のあるもののみカバーした。
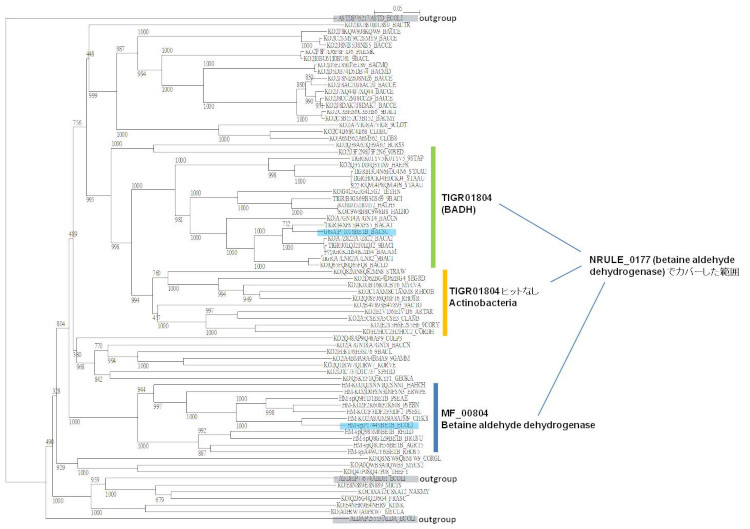
Phylogenetic tree of betaine aldehyde dehydrogenase -
2ステップでベタインを合成する経路 (4)
コリンをベタインに変換するcholine oxidase (EC 1.1.3.17 , NRULE_0178) は放線菌の Arthrobacter globiformis で実験されている。
この酵素に特異的なタンパク質モチーフはなく、構造は BetA と同じ glucose-methanol-choline oxidoreductase family のメンバーである。
, NRULE_0178) は放線菌の Arthrobacter globiformis で実験されている。
この酵素に特異的なタンパク質モチーフはなく、構造は BetA と同じ glucose-methanol-choline oxidoreductase family のメンバーである。
グリシンからベタインを生合成する経路 (NFUNC_0050)
グリシンの 3メチル化によりベタインを合成する経路には、2つのメチル化酵素による経路と、1つの酵素でトリメチル化する経路がある。ベタイン合成に関わる細菌のメチル化酵素は基質特異性により 4つに分けられる。
-
2つのメチル化酵素による経路 (5) (6) (7)
グリシンからジメチルグリシンまで触媒する glycine/sarcosine methytransferase EC 2.1.1.156 , NRULE_0179
には固有のタンパク質モチーフがあるが (PIRSF000385
, NRULE_0179
には固有のタンパク質モチーフがあるが (PIRSF000385 )、次のステップを触媒する sarcosine/dimethylglycine methyltransferase (EC 2.1.1.157
)、次のステップを触媒する sarcosine/dimethylglycine methyltransferase (EC 2.1.1.157 )、
dimethylglycine methyltransferase (EC 2.1.1.161
)、
dimethylglycine methyltransferase (EC 2.1.1.161 ) は一般的な methyltransferase ドメインしか持っていない。
この2つは相同性があり、BLASTP の結果からも区別することが難しいため MiFuP では、この 2つの酵素を区別せずに、
EC 2.1.1.161
) は一般的な methyltransferase ドメインしか持っていない。
この2つは相同性があり、BLASTP の結果からも区別することが難しいため MiFuP では、この 2つの酵素を区別せずに、
EC 2.1.1.161 としてルール化した (NRULE_0180)。
詳細については 図、ベタイン合成に関わる細菌のメチル化酵素参照。
としてルール化した (NRULE_0180)。
詳細については 図、ベタイン合成に関わる細菌のメチル化酵素参照。
-
1つのメチル化酵素による経路 (8)
glycine/sarcosine/dimethylglycine methyltransferase (EC 2.1.1.156 /EC 2.1.1.157
/EC 2.1.1.157 )は Actinopolyspora halophila で実験されている。
一般的な methyltransferase 構造で、EC 2.1.1.157
)は Actinopolyspora halophila で実験されている。
一般的な methyltransferase 構造で、EC 2.1.1.157 および EC 2.1.1.161
および EC 2.1.1.161 と相同性を示すが、BLASTP の結果からこれらと判別することは可能である (NRULE_0181)。
と相同性を示すが、BLASTP の結果からこれらと判別することは可能である (NRULE_0181)。
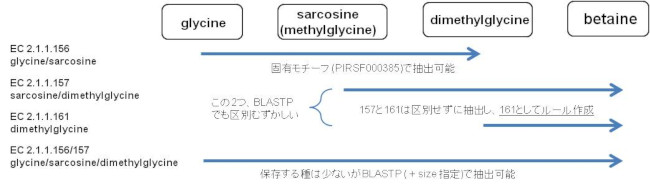
ベタイン合成に関わる細菌のメチル化酵素
機能を持つことが知られている生物
コリンから合成する経路は、プロテオバクテリアの Escherichia coli や Chromohalobacter salexigens、
グリシンを出発物質とする合成経路については放線菌の Aphanothece halophytica などで活性を持つことが実験で確認されている。
実用化例
双性イオン、アミノ酸、およびコリン代謝物それぞれの特性を生かして産業利用されている。
- 食品添加物 (調味料) としての使用
- 保湿剤として化粧品やシャンプーなどへの添加
- ホモシスチン尿症の処方薬
生体内では、コリンの主な代謝産物でメチル基供与体としてホモシステインからメチオニンへの再メチル化に関与することから、ホモシスチン尿症(Cystathionine beta-synthase が先天的に欠損し、 血中メチオニン濃度が上昇)の処方薬として承認されている。
参考文献
- 仲山英樹 (2012) 「好塩菌の塩ストレス適 応機構とその応用」『生物工学会誌』No. 98-11, pp696-700.
- Landfald, B. and Strom, A. R. (1986). Choline-glycine betaine pathway confers a high level of osmotic tolerance in Escherichia coli. J Bacteriol. 165(3):849-855. PMID: 3512525

- Gadda, G. and McAllister-Wilkins, E. E. (2003). Cloning, expression, and purification of choline dehydrogenase from the moderate halophile Halomonas elongata. Appl Environ Microbiol. 69(4):2126-2132. PMID: 12676692

- Roberts, M. F. (2005). Organic compatible solutes of halotolerant and halophilic microorganisms. Saline Systems. 1:5. PMID: 16176595

関連外部リンク
- MetaCyc Pathway: Betaine-Biosynthesis

MiFuPへのリンク
- NFUNC_0049 Betaine biosynthesis (from choline)
NRULE_0175 Choline dehydrogenase
NRULE_0176 Alcohol dehydrogenase
NRULE_0177 Betaine aldehyde dehydrogenase
NRULE_0178 Choline oxidase - NFUNC_0050 Betaine biosynthesis (from glycine)
NRULE_0179 Glycine/sarcosine N-methyltransferase
NRULE_0180 Dimethylglycine N-methyltransferase
NRULE_0181 Glycine/sarcosine/dimethylglycine N-methyltransferase
(更新日 2014/03/12)