Pikro_00050 : CDS information
 Location
Location
| Organism | Streptomyces venezuelae |
|---|---|
| Strain | ATCC 15439 |
| Entry name | Pikromycin |
| Contig | AF079138 |
| Start / Stop / Direction | 28,161 / 32,849 / + [in whole cluster] 28,161 / 32,849 / + [in contig] |
| Location | 28161..32849 [in whole cluster] 28161..32849 [in contig] |
| Type | CDS |
| Length | 4,689 bp (1,562 aa) |
 Annotation
Annotation
| Category | 1.1 PKS |
|---|---|
| Product | polyketide synthase |
| Product (GenBank) | type I polyketide synthase PikAIII |
| Gene | |
| Gene (GenBank) | pikAIII |
| EC number | |
| Keyword | |
| Note | |
| Note (GenBank) |
|
| Reference |
|
 PKS/NRPS Module
PKS/NRPS Module
| 5 | 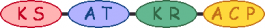 | methylmalonyl-CoA |
 Sequence
Sequence
>polyketide synthase [type I polyketide synthase PikAIII] MANNEDKLRDYLKRVTAELQQNTRRLREIEGRTHEPVAIVGMACRLPGGVASPEDLWQLV AGDGDAISEFPQDRGWDVEGLYDPDPDASGRTYCRSGGFLHDAGEFDADFFGISPREALA MDPQQRLSLTTAWEAIESAGIDPTALKGSGLGVFVGGWHTGYTSGQTTAVQSPELEGHLV SGAALGFLSGRIAYVLGTDGPALTVDTACSSSLVALHLAVQALRKGECDMALAGGVTVMP NADLFVQFSRQRGLAADGRSKAFATSADGFGPAEGAGVLLVERLSDARRNGHRILAVVRG SAVNQDGASNGLTAPHGPSQQRVIRRALADARLAPGDVDVVEAHGTGTRLGDPIEAQALI ATYGQEKSSEQPLRLGALKSNIGHTQAAAGVAGVIKMVQAMRHGLLPKTLHVDEPSDQID WSAGTVELLTEAVDWPEKQDGGLRRAAVSSFGISGTNAHVVLEEAPAVEDSPAVEPPAGG GVVPWPVSAKTPAALDAQIGQLAAYADGRTDVDPAVAARALVDSRTAMEHRAVAVGDSRE ALRDALRMPEGLVRGTSSDVGRVAFVFPGQGTQWAGMGAELLDSSPEFAASMAECETALS RYVDWSLEAVVRQEPGAPTLDRVDVVQPVTFAVMVSLAKVWQHHGITPQAVVGHSQGEIA AAYVAGALTLDDAARVVTLRSKSIAAHLAGKGGMISLALDEAAVLKRLSDFDGLSVAAVN GPTATVVSGDPTQIEELARTCEADGVRARIIPVDYASHSRQVEIIEKELAEVLAGLAPQA PHVPFFSTLEGTWITEPVLDGTYWYRNLRHRVGFAPAVETLAVDGFTHFIEVSAHPVLTM TLPETVTGLGTLRREQGGQERLVTSLAEAWANGLTIDWAPILPTATGHHPELPTYAFQTE RFWLQSSAPTSAADDWRYRVEWKPLTASGQADLSGRWIVAVGSEPEAELLGALKAAGAEV DVLEAGADDDREALAARLTALTTGDGFTGVVSLLDDLVPQVAWVQALGDAGIKAPLWSVT QGAVSVGRLDTPADPDRAMLWGLGRVVALEHPERWAGLVDLPAQPDAAALAHLVTALSGA TGEDQIAIRTTGLHARRLARAPLHGRRPTRDWQPHGTVLITGGTGALGSHAARWMAHHGA EHLLLVSRSGEQAPGATQLTAELTASGARVTIAACDVADPHAMRTLLDAIPAETPLTAVV HTAGAPGGDPLDVTGPEDIARILGAKTSGAEVLDDLLRGTPLDAFVLYSSNAGVWGSGSQ GVYAAANAHLDALAARRRARGETATSVAWGLWAGDGMGRGADDAYWQRRGIRPMSPDRAL DELAKALSHDETFVAVADVDWERFAPAFTVSRPSLLLDGVPEARQALAAPVGAPAPGDAA VAPTGQSSALAAITALPEPERRPALLTLVRTHAAAVLGHSSPDRVAPGRAFTELGFDSLT AVQLRNQLSTVVGNRLPATTVFDHPTPAALAAHLHEAYLAPAEPAPTDWEGRVRRALAEL PLDRLRDAGVLDTVLRLTGIEPEPGSGGSDGGAADPGAEPEASIDDLDAEALIRMALGPR NT
>polyketide synthase [type I polyketide synthase PikAIII] ATGGCGAACAACGAAGACAAGCTCCGCGACTACCTCAAGCGCGTCACCGCCGAGCTGCAG CAGAACACCAGGCGTCTGCGCGAGATCGAGGGACGCACGCACGAGCCGGTGGCGATCGTG GGCATGGCCTGCCGCCTGCCGGGCGGTGTCGCCTCGCCCGAGGACCTGTGGCAGCTGGTG GCCGGGGACGGGGACGCGATCTCGGAGTTCCCGCAGGACCGCGGCTGGGACGTGGAGGGG CTGTACGACCCCGACCCGGACGCGTCCGGCAGGACGTACTGCCGGTCCGGCGGATTCCTG CACGACGCCGGCGAGTTCGACGCCGACTTCTTCGGGATCTCGCCGCGCGAGGCCCTCGCC ATGGACCCGCAGCAGCGACTGTCCCTCACCACCGCGTGGGAGGCGATCGAGAGCGCGGGC ATCGACCCGACGGCCCTGAAGGGCAGCGGCCTCGGCGTCTTCGTCGGCGGCTGGCACACC GGCTACACCTCGGGGCAGACCACCGCCGTGCAGTCGCCCGAGCTGGAGGGCCACCTGGTC AGCGGCGCGGCGCTGGGCTTCCTGTCCGGCCGTATCGCGTACGTCCTCGGTACGGACGGA CCGGCCCTGACCGTGGACACGGCCTGCTCGTCCTCGCTGGTCGCCCTGCACCTCGCCGTG CAGGCCCTCCGCAAGGGCGAGTGCGACATGGCCCTCGCCGGTGGTGTCACGGTCATGCCC AACGCGGACCTGTTCGTGCAGTTCAGCCGGCAGCGCGGGCTGGCCGCGGACGGCCGGTCG AAGGCGTTCGCCACCTCGGCGGACGGCTTCGGCCCCGCGGAGGGCGCCGGAGTCCTGCTG GTGGAGCGCCTGTCGGACGCCCGCCGCAACGGACACCGGATCCTCGCGGTCGTCCGCGGC AGCGCGGTCAACCAGGACGGCGCCAGCAACGGCCTCACGGCTCCGCACGGGCCCTCCCAG CAGCGCGTCATCCGACGGGCCCTGGCGGACGCCCGGCTCGCGCCGGGTGACGTGGACGTC GTCGAGGCGCACGGCACGGGCACGCGGCTCGGCGACCCGATCGAGGCGCAGGCCCTCATC GCCACCTACGGCCAGGAGAAGAGCAGCGAACAGCCGCTGAGGCTGGGCGCGTTGAAGTCG AACATCGGGCACACGCAGGCCGCGGCCGGTGTCGCAGGTGTCATCAAGATGGTCCAGGCG ATGCGCCACGGACTGCTGCCGAAGACGCTGCACGTCGACGAGCCCTCGGACCAGATCGAC TGGTCGGCGGGCACGGTGGAACTCCTCACCGAGGCCGTCGACTGGCCGGAGAAGCAGGAC GGCGGGCTGCGCCGCGCGGCTGTCTCCTCCTTCGGCATCAGCGGGACGAACGCGCACGTC GTCCTGGAGGAGGCCCCGGCGGTCGAGGACTCCCCGGCCGTCGAGCCGCCGGCCGGTGGC GGTGTGGTGCCGTGGCCGGTGTCCGCGAAGACTCCGGCCGCGCTGGACGCCCAGATCGGG CAGCTCGCCGCGTACGCGGACGGTCGTACGGACGTGGATCCGGCGGTGGCCGCCCGCGCC CTGGTCGACAGCCGTACGGCGATGGAGCACCGCGCGGTCGCGGTCGGCGACAGCCGGGAG GCACTGCGGGACGCCCTGCGGATGCCGGAAGGACTGGTACGCGGCACGTCCTCGGACGTG GGCCGGGTGGCGTTCGTCTTCCCCGGCCAGGGCACGCAGTGGGCCGGCATGGGCGCCGAA CTCCTTGACAGCTCACCGGAGTTCGCTGCCTCGATGGCCGAATGCGAGACCGCGCTCTCC CGCTACGTCGACTGGTCTCTTGAAGCCGTCGTCCGACAGGAACCCGGCGCACCCACGCTC GACCGCGTCGACGTCGTCCAGCCCGTGACCTTCGCTGTCATGGTCTCGCTGGCGAAGGTC TGGCAGCACCACGGCATCACCCCCCAGGCCGTCGTCGGCCACTCGCAGGGCGAGATCGCC GCCGCGTACGTCGCCGGTGCACTCACCCTCGACGACGCCGCCCGCGTCGTCACCCTGCGC AGCAAGTCCATCGCCGCCCACCTCGCCGGCAAGGGCGGCATGATCTCCCTCGCCCTCGAC GAGGCGGCCGTCCTGAAGCGACTGAGCGACTTCGACGGACTCTCCGTCGCCGCCGTCAAC GGCCCCACCGCCACCGTCGTCTCCGGCGACCCGACCCAGATCGAGGAACTCGCCCGCACC TGCGAGGCCGACGGCGTCCGTGCGCGGATCATCCCGGTCGACTACGCCTCCCACAGCCGG CAGGTCGAGATCATCGAGAAGGAGCTGGCCGAGGTCCTCGCCGGACTCGCCCCGCAGGCT CCGCACGTGCCGTTCTTCTCCACCCTCGAAGGCACCTGGATCACCGAGCCGGTGCTCGAC GGCACCTACTGGTACCGCAACCTGCGCCATCGCGTGGGCTTCGCCCCCGCCGTGGAGACC TTGGCGGTTGACGGCTTCACCCACTTCATCGAGGTCAGCGCCCACCCCGTCCTCACCATG ACCCTCCCCGAGACCGTCACCGGCCTCGGCACCCTCCGCCGCGAACAGGGAGGCCAGGAG CGTCTGGTCACCTCACTCGCCGAAGCCTGGGCCAACGGCCTCACCATCGACTGGGCGCCC ATCCTCCCCACCGCAACCGGCCACCACCCCGAGCTCCCCACCTACGCCTTCCAGACCGAG CGCTTCTGGCTGCAGAGCTCCGCGCCCACCAGCGCCGCCGACGACTGGCGTTACCGCGTC GAGTGGAAGCCGCTGACGGCCTCCGGCCAGGCGGACCTGTCCGGGCGGTGGATCGTCGCC GTCGGGAGCGAGCCAGAAGCCGAGCTGCTGGGCGCGCTGAAGGCCGCGGGAGCGGAGGTC GACGTACTGGAAGCCGGGGCGGACGACGACCGTGAGGCCCTCGCCGCCCGGCTCACCGCA CTGACGACCGGCGACGGCTTCACCGGCGTGGTCTCGCTCCTCGACGACCTCGTGCCACAG GTCGCCTGGGTGCAGGCACTCGGCGACGCCGGAATCAAGGCGCCCCTGTGGTCCGTCACC CAGGGCGCGGTCTCCGTCGGACGTCTCGACACCCCCGCCGACCCCGACCGGGCCATGCTC TGGGGCCTCGGCCGCGTCGTCGCCCTTGAGCACCCCGAACGCTGGGCCGGCCTCGTCGAC CTCCCCGCCCAGCCCGATGCCGCCGCCCTCGCCCACCTCGTCACCGCACTCTCCGGCGCC ACCGGCGAGGACCAGATCGCCATCCGCACCACCGGACTCCACGCCCGCCGCCTCGCCCGC GCACCCCTCCACGGACGTCGGCCCACCCGCGACTGGCAGCCCCACGGCACCGTCCTCATC ACCGGCGGCACCGGAGCCCTCGGCAGCCACGCCGCACGCTGGATGGCCCACCACGGAGCC GAACACCTCCTCCTCGTCAGCCGCAGCGGCGAACAAGCCCCCGGAGCCACCCAACTCACC GCCGAACTCACCGCATCGGGCGCCCGCGTCACCATCGCCGCCTGCGACGTCGCCGACCCC CACGCCATGCGCACCCTCCTCGACGCCATCCCCGCCGAGACGCCCCTCACCGCCGTCGTC CACACCGCCGGCGCACCGGGCGGCGATCCGCTGGACGTCACCGGCCCGGAGGACATCGCC CGCATCCTGGGCGCGAAGACGAGCGGCGCCGAGGTCCTCGACGACCTGCTCCGCGGCACT CCGCTGGACGCCTTCGTCCTCTACTCCTCGAACGCCGGGGTCTGGGGCAGCGGCAGCCAG GGCGTCTACGCGGCGGCCAACGCCCACCTCGACGCGCTCGCCGCCCGGCGCCGCGCCCGG GGCGAGACGGCGACCTCGGTCGCCTGGGGCCTCTGGGCCGGCGACGGCATGGGCCGGGGC GCCGACGACGCGTACTGGCAGCGTCGCGGCATCCGTCCGATGAGCCCCGACCGCGCCCTG GACGAACTGGCCAAGGCCCTGAGCCACGACGAGACCTTCGTCGCCGTGGCCGATGTCGAC TGGGAGCGGTTCGCGCCCGCGTTCACGGTGTCCCGTCCCAGCCTTCTGCTCGACGGCGTC CCGGAGGCCCGGCAGGCGCTCGCCGCACCCGTCGGTGCCCCGGCTCCCGGCGACGCCGCC GTGGCGCCGACCGGGCAGTCGTCGGCGCTGGCCGCGATCACCGCGCTCCCCGAGCCCGAG CGCCGGCCGGCGCTCCTCACCCTCGTCCGTACCCACGCGGCGGCCGTACTCGGCCATTCC TCCCCCGACCGGGTGGCCCCCGGCCGTGCCTTCACCGAGCTCGGCTTCGACTCGCTGACG GCCGTGCAGCTCCGCAACCAGCTCTCCACGGTGGTCGGCAACAGGCTCCCCGCCACCACG GTCTTCGACCACCCGACGCCCGCCGCACTCGCCGCGCACCTCCACGAGGCGTACCTCGCA CCGGCCGAGCCGGCCCCGACGGACTGGGAGGGGCGGGTGCGCCGGGCCCTGGCCGAACTG CCCCTCGACCGGCTGCGGGACGCGGGGGTCCTCGACACCGTCCTGCGCCTCACCGGCATC GAGCCCGAGCCGGGTTCCGGCGGTTCGGACGGCGGCGCCGCCGACCCTGGTGCGGAGCCG GAGGCGTCGATCGACGACCTGGACGCCGAGGCCCTGATCCGGATGGCTCTCGGCCCCCGT AACACCTGA
 Feature
Feature
| BLASTP Database:UniProtKB:2011_09 |
|
|---|---|
| InterPro Database:interpro:38.0 |
IPR001227 Acyl transferase domain (Domain)
IPR002198 Short-chain dehydrogenase/reductase SDR (Family)
IPR006162 Phosphopantetheine attachment site (PTM)
IPR009081 Acyl carrier protein-like (Domain)
IPR014030 Beta-ketoacyl synthase, N-terminal (Domain)
IPR014031 Beta-ketoacyl synthase, C-terminal (Domain)
IPR014043 Acyl transferase (Domain)
IPR015083 Polyketide synthase, docking (Domain)
IPR016035 Acyl transferase/acyl hydrolase/lysophospholipase (Domain)
IPR016036 Malonyl-CoA ACP transacylase, ACP-binding (Domain)
IPR016038 Thiolase-like, subgroup (Domain)
IPR016039 Thiolase-like (Domain)
IPR016040 NAD(P)-binding domain (Domain)
IPR018201 Beta-ketoacyl synthase, active site (Active_site)
IPR020801 Polyketide synthase, acyl transferase domain (Domain)
IPR020806 Polyketide synthase, phosphopantetheine-binding domain (Domain)
IPR020841 Polyketide synthase, beta-ketoacyl synthase domain (Domain)
IPR020842 Polyketide synthase/Fatty acid synthase, KR (Domain)
|
| SignalP | No significant hit |
| TMHMM | No significant hit |


 Pikro_00040
Pikro_00040


