利用菌株の安全性確認
Acinetobacter属
・ Multilocus sequence analysis (MLSA)法の開発・ MLSA法手順書の入手
・ PCR法によるAcinetobacter baumaniiの簡便検出法
Comamonas属
・ MLSA法の開発
これらの手法は岐阜大学大学院 江崎孝行教授の研究グループと共同で開発しました
Acinetobacter属
Acinetobacter属細菌は土壌や河川水などの自然環境中に広く生息する細菌で、 環境汚染物質である3, 4-ジクロロアニリン分解菌(NBRC 109690)や原油分解菌(NBRC 108641、NBRC 100985)など私たちにとって有用な菌株が存在し、 これまでに41の菌種が知られています(2016年3月現在)。 しかし、 Acinetobacter属細菌にはAcinetobacter baumanniiやAcinetobacter lwoffiiをはじめ、いくつかのヒト病原菌種も存在しています。 また、近年では抗生物質が効かない多剤耐性のAcinetobacter属細菌が世界的に問題となっており、日本でも広域β-ラクタム剤、アミノ配糖体、フルオロキノロンの3系統の薬剤に耐性を示すAcinetobacter属細菌による感染症は、2011年に感染症法で定める5類感染症に加えられ、定点把握の対象となっています。
Acinetobacter属細菌は各菌種間における16S rRNA遺伝子配列の多型が少なく、正確な分類ができないことが問題となっています。 (Kampfer & Glaeser 2011, Microbiology Australia, 32: 66【PDF:201KB】; Chan et al. 2012, BMC Microbiology, 12: 302)。このことから、Acinetobacter属の基準株15株について全ゲノム解析を実施し、 既にゲノム配列が公開されている基準株と合わせ、現時点で解析可能な全28種についてMLSA法の開発を実施しました。
Multilocus sequence analysis (MLSA)法の開発
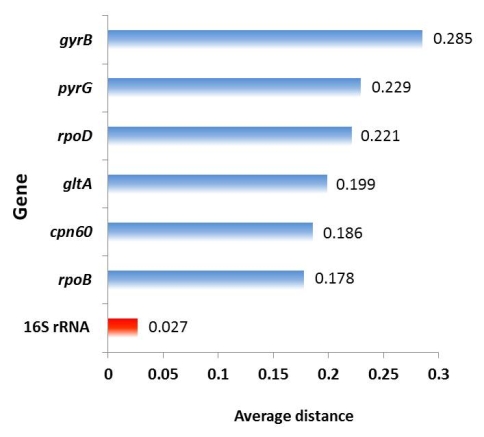
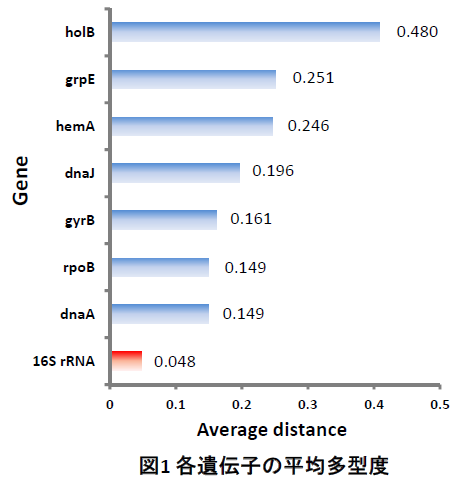
A. baumanniiのMLST(Multilocus sequence typing)*1, 2 で用いられ、28株に共通してシングルコピーであった6個のハウスキーピング遺伝子の塩基配列を使用しました。使用した遺伝子の平均多型は16S rRNA遺伝子(1225 bp)に比べて大きいことがわかります(図1)。
(クリックして画像を拡大)

*1. ハウスキーピング遺伝子配列に基づいて菌株のタイピングを行い、菌株を識別する方法。
*2. PubMLST website (http://pubmlst.org/) オックスフォード大学が運営しているデータベース( Jolley & Maiden 2010, BMC Bioinformatics, 11:595 )
6種類のハウスキーピング遺伝子 (cpn60, gltA, gyrB, pyrG, rpoB, rpoD) を連結した塩基配列を用いてMLSAを行った結果、16S rRNA遺伝子の塩基配列で描いた系統樹に比べて信頼性を示すブートストラップ値(系統樹上の数字)が飛躍的に向上し、近縁関係がより明確になりました(図2)。
MLSA法手順書の入手
| 様式 | 内容 | 最終更新日 | ダウンロード |
|---|---|---|---|
| MLSA法によるAcinetobacter属細菌の 同定法 |
全ゲノム配列からのハウスキーピング遺伝子配列の取得から、分子系統解析までの手順書です。 | 2016年3月25日 | MLSA法手順書 【PDF:4.01MB】 |
PCR法によるAcinetobacter baumanniiの簡便検出法
Acinetobacter baumanniiは医療機関において最も多く検出されるAcinetobacter属細菌です。しかし、Acinetobacter nosocomialis、Acinetobacter pittii 、Acinetobacter calcoaceticusとは表現型が非常に類似していることから、医療機関で用いられている表現性状に基づく簡易同定法では正確に鑑別することができず、これら3菌種もA. baumanniiと誤って判定されてしまうケースがかなりあります。このことから、医療機関においてA. baumanniiの正確な分離症例数を把握することは困難となっています。そこで、A. baumanniiのみを簡便に検出するために、gyrB、rpoB、recAなど11個のハウスキーピング遺伝子についてPCRプライマーを設計し、Acinetobacter基準株29株に対する特異性を調べました。その結果、A. baumanniiの recA遺伝子のみを特異的に増幅するPCRプライマーの設計に成功しました。
手順
表1にプライマーの配列を、表2にPCRの反応液組成と反応条件を示しました。PCR終了後、電気泳動により増幅を確認しました。本実験では、Biometra TProfessional Basic 96 thermocycler (BM Equipment Co.,Ltd.) を使用しました。

検出例1
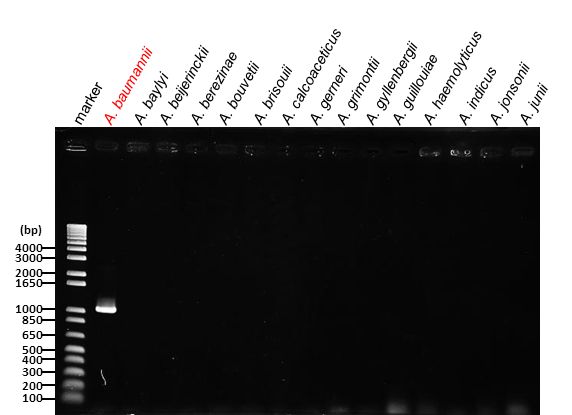
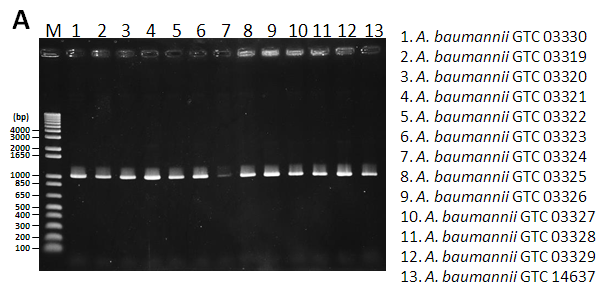
開発したプライマー(recAFとrecAR) の特異性を調べるために、Acinetobacter属の基準株29株についてPCRを行いました。その結果、A. baumanniiのみrecAの増幅が観察されました(図1 赤色の矢印で示したバンド)。
図1 Acinetobacter基準株でのPCR結果 (クリックして画像を拡大)
検出例2
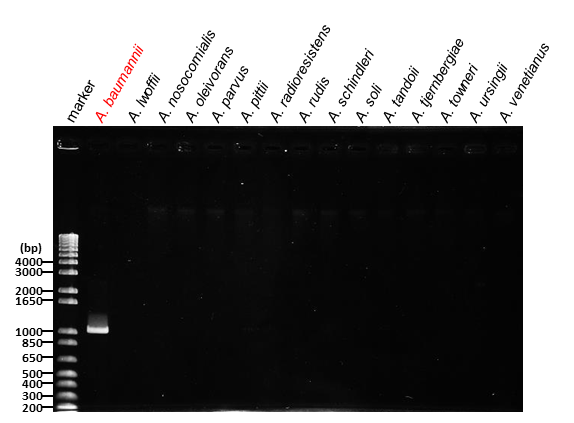
基準株以外の Acinetobacter属34株に対してPCRを行い、 開発したプライマーの特異性を検証しました。その結果、A. baumanniiのみrecAの増幅が観察されました。
 |
|
 |
 |
図2 Acinetobacter臨床株でのPCR結果 (クリックして画像を拡大)
(M: marker)
Comamonas属
Comamonas属は、土壌や地下水等の自然環境中に広く生息する細菌です。 これまでに約17の菌種が知られており、Comamonas aquatica、Comamonas kerstersii、Comamonas terrigena、Comamonas testosteroniはまれに臨床検体からも単離されることから、 日本細菌学会における病原細菌のバイオセーフティレベル(BSL)で日和見病原体(BSL1*)に分類されています。Comamonas属はおおむね16S rRNA遺伝子配列に基づいた分類が可能ですが、 日和見病原体に該当するC. testosteroniと非病原体であるComamonas thiooxidansでは16S rRNA遺伝子配列が100%一致しており区別できないという問題があります。 また、C. testosteroni-C. thiooxidansのグループには、 4-塩素化ニトロベンゼン、ビフェニル、フタル酸および原油等の分解菌の報告があり、これらの産業有用株の安全性を評価する上でC. testosteroni-C. thiooxidansの判別が重要です。 そこで、Comamonas属の基準株17株について全ゲノム解析を実施し、MLSAの開発を行いました。
MLSA法の開発
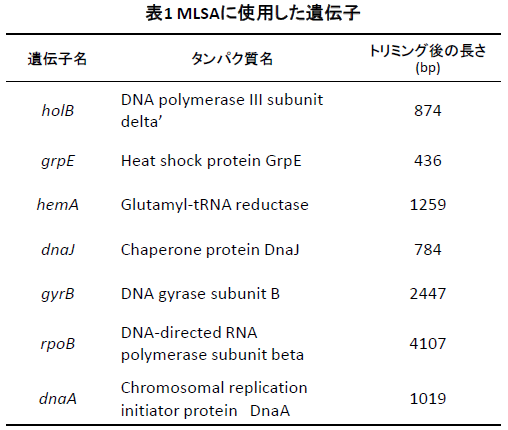
Comamonas属およびその類縁な属で系統解析に利用されている表1に示した7種類のハウスキーピング遺伝子を用いました。
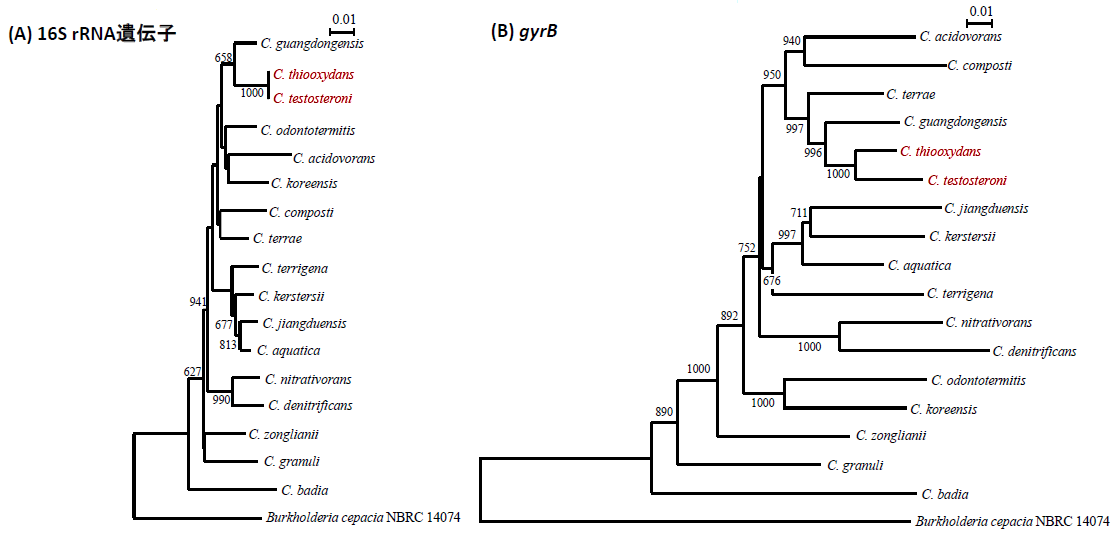
7種類のハウスキーピング遺伝子 ( dnaA, grpE, hemA, rpoB, dnaJ, gyrB, holB) を連結した塩基配列を用いてMLSAを行った結果、 16S rRNA遺伝子の塩基配列で描いた系統樹に比べて信頼性を示すブートストラップ値(系統樹上の数字)が飛躍的に向上しました。 また、 C. testosteroniとC. thiooxidansを明確に区別することができました。
PCR法によるComamonas属のGyrBユニバーサルプライマーの開発
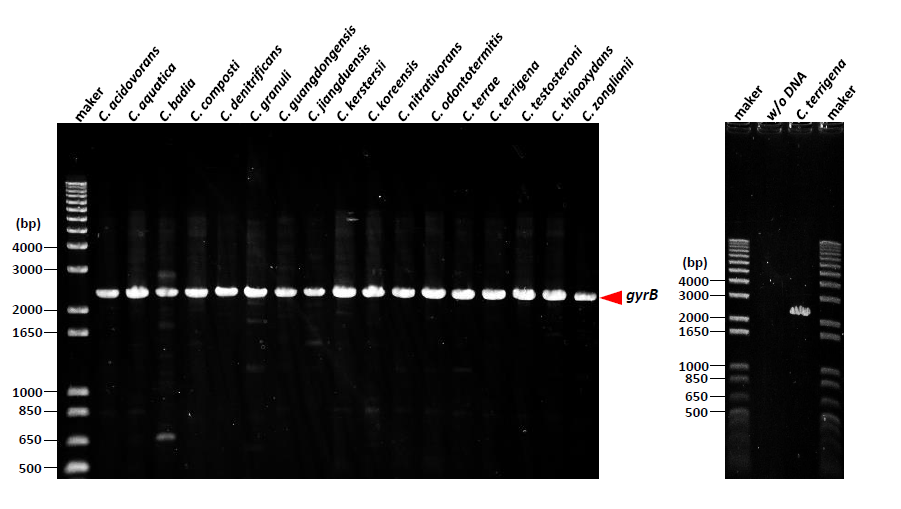
バイオレメディエーションに微生物を利用するためには、利用微生物の属種名を同定する必要があります。 バイオレメディエーション利用指針の解説*1では「16S rRNA 遺伝子の98.5%以上のホモロジーを持つ場合に同種とみなす。 なお、98.5%以上のホモロジーを持つものの中に複数の独立種が存在する場合は、全ゲノム遺伝子の解析又はジャイレースB 遺伝子(GyrB)等の解析若しくは表現型を中心とした手法により同定する。」とされています。 Comamonas属ではC. testosteroniとC. thiooxidansの16S rRNA遺伝子配列が100%一致しており、区別することができません。一方、GyrB遺伝子配列に基づいた系統解析では、 16S rRNA 遺伝子に比べて解像度が高く、 C. testosteroniとC. thiooxidansも明確に区別することができます(図1)。そこで、 Comamonas属を明確に分類するためにGyrB遺伝子を増幅するユニバーサルプライマーを開発しました。
- *1. 微生物によるバイオレメディエーション利用指針の解説 参考URL: (経済産業省のホームページ)
- http://www.meti.go.jp/policy/mono_info_service/mono/bio/cartagena/baireme_kaisetsu.pdf【PDF:493KB】
手順
表1にユニバーサルプライマーの配列を、表2にPCRの反応液組成と反応条件を示しました。PCR終了後、電気泳動により増幅を確認しました。さらに表2に示したシーケンスプライマーを用いて直接シーケンスを行い、gyrB全長をシーケンスできることも確認しました。


結果
Comamonas属の全基準株17株についてPCRを行い、全ての基準株でgyrBのほぼ全長に相当する2.4 kbの増幅バンドが観察されました(図2 赤色の矢印で示したバンド)。表3のプライマーを用いてシーケンスし、増幅したgyrB断片の配列を得ることができました。
PDFファイルをご覧いただくためには、Adobe Reader(無償)が必要です。Adobe Readerはダウンロードページ![]() よりダウンロードできます。
よりダウンロードできます。
お問い合わせ
- 独立行政法人製品評価技術基盤機構 バイオテクノロジーセンター バイオ技術評価・開発課(東京)
-
TEL:03-3481-1936
住所:〒151-0066 東京都渋谷区西原2-49-10 地図
お問い合わせフォームへ